科研进展
Nature丨王程远等解析细菌Rho因子依赖型转录终止的结构基础
发表日期: 2023-01-30
1月25日,美国罗格斯大学 Richard Ebright 团队在Nature上发表题为Structural basis of Rho-dependent transcription termination的研究论文,该研究解析了细菌Rho因子依赖型转录终止复合体冷冻电镜结构,揭示了细菌Rho因子介导RNA聚合酶转录终止的分子机制。本文的共同第一作者及共同通讯作者王程远博士现为研究所微生物、发育与健康研究中心研究员。

Rho因子依赖型转录终止(Rho dependent transcription termination)是普遍存在于细菌中的一类转录终止机制。与终止信号编码在DNA中的固有终止不同,Rho因子依赖型转录终止主要通过具有5′ 3′转移酶/解旋酶活性的Rho因子完成RNA聚合酶转录终止过程,同时与转录翻译偶联过程相互协作,在细菌应激基因表达、外源基因沉默、毒力因子调控、维持基因组稳定性等过程起着重要调控作用。
细菌Rho因子依赖型转录终止过程主要包括四个步骤:1)Rho解旋酶形成开环状态(open-ring state)六聚体,并通过其初始RNA结合位点(Primary binding site,PBS)识别信使RNA上的特殊序列(Rho utilization site,rut site)并与信使RNA结合;2)开环状态Rho六聚体通过其中心位置第二个RNA结合位点(secondary binding site,SBS)与富嘧啶RNA序列结合;3)开环状态六聚体闭合形成闭环六聚体(close-ring state),并通过水解ATP提供能量沿着5’-3’方向在信使RNA上移动;4)Rho解旋酶六聚体接近延伸状态RNA聚合酶,并通过NusG转录因子介导与RNA聚合酶结合,通过水解ATP提供机械力解离信使RNA与RNA聚合酶,最终完成RNA聚合酶的转录终止过程。
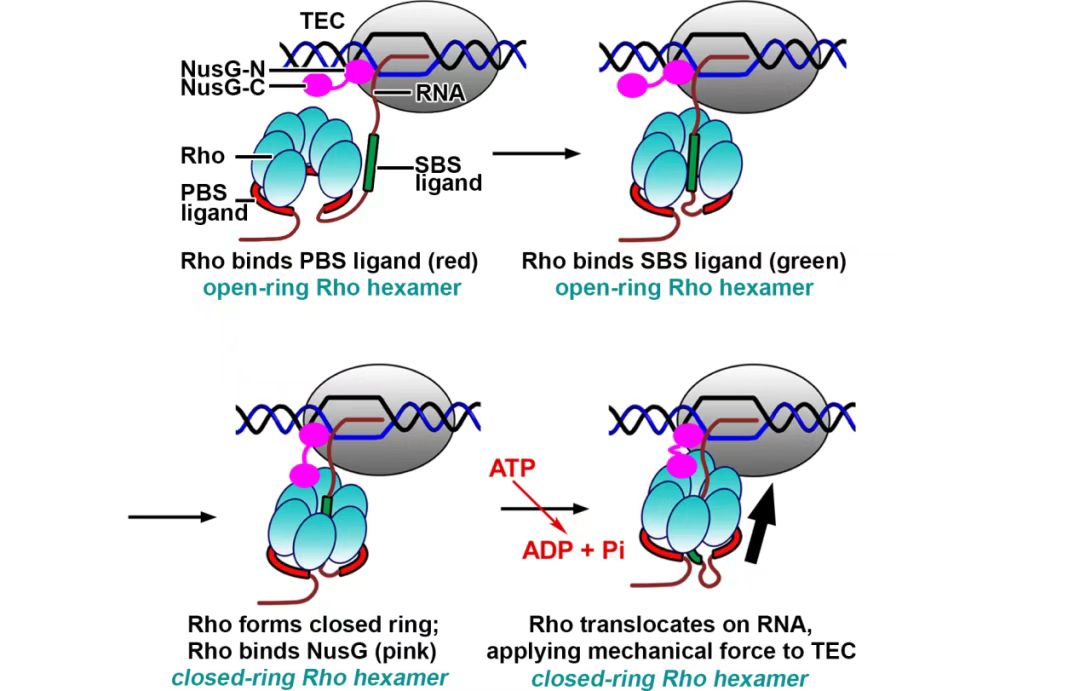
图1:Rho依赖型转录终止过程模型图
然而Rho因子如何识别rut序列、NusG如何介导RNA聚合酶与Rho的相互作用等机制尚不清晰。研究团队首先设计带有不同长度信使RNA的核酸骨架,测试不同组合情况下(不同PBS底物 tR1、dC5、dC15和dC75;NusG;ATP等)体外转录终止效率,确定Rho依赖的转录终止需要PBS底物( tR1、dC75)、NusG、ATP、Rho同时存在的情况下才能发挥转录终止活性,并且当信使RNA spacer为6密码子长度时具有最高的终止效率;之后通过单颗粒冷冻电镜方法分别解析了Rho-NusG-RNAP与不同PBS底物结合的转录终止复合体结构(分辨率为4.2-6.5 )。最终基于这些结构,研究团队证明:
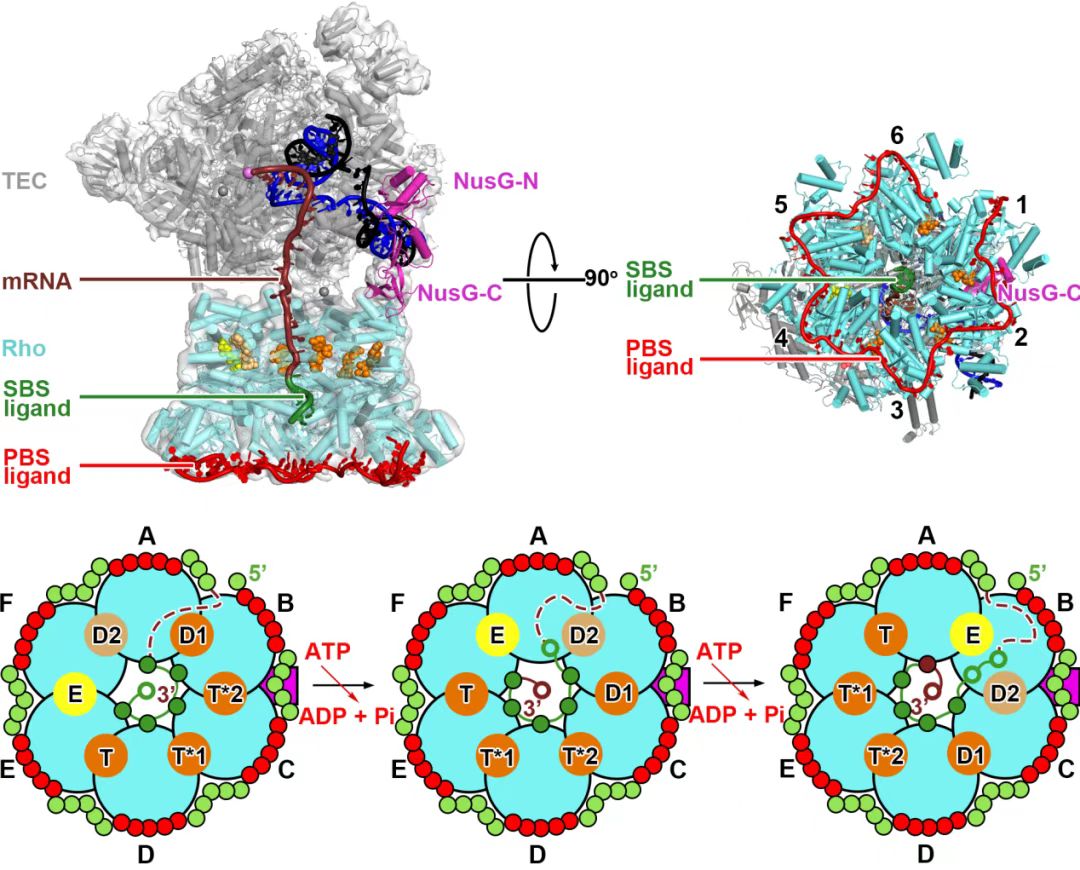
图2:转录终止复合体结构(上)及Rho因子驱动模型(下)
1)NusG蛋白通过N端结构域结合RNA聚合酶,C端KOW结构域结合Rho蛋白C端结构域,连接RNA聚合酶和Rho因子;
2)Rho因子六聚体处于闭环状态,其PBS位点特异性与信使RNA rut序列结合,SBS位点与信使RNA spacer部分结合;
3)Rho因子六聚体分别结合ATP分子,依次连续发生水解反应,提供能量驱动Rho因子沿着5′ 3′方向在RNA上移动,并最终通过对RNA聚合酶施加机械力,破坏其活性反应中心RNA-DNA杂合双链,完成转录终止过程;
4)RNA聚合酶通过 亚基、 亚基FTH结构域、信使RNA以及NusG与Rho因子相互作用,最终完成转录终止反应。
该研究报道了细菌Rho依赖型转录终止复合体冷冻电镜结构,解析了Rho因子特异性识别rut序列,PBS、SBS位点分别结合信使RNA的分子机制,揭示了NusG蛋白介导Rho因子与RNA聚合酶结合的作用方式。该研究回答了转录终止的关键基础科学问题。
附件下载: