Journal of Cell Biology|酒亚明课题组发现细胞迁移模式调控迁移体形成的新现象
2月18日,中国科学院上海巴斯德研究所酒亚明课题组在Journal of Cell Biology上在线发表了题为“Cell migration orchestrates migrasome formation by shaping retraction fibers”的文章,该研究通过定量分析细胞迁移模式的两个关键指标(持续性persistence和速度velocity)对迁移体形成的调控,发现细胞持续性可以通过影响收缩纤维进而影响迁移体的形成。

迁移体(migrasome)是最近发现的一种具有囊泡状形态的细胞结构。当细胞在细胞外基质上爬行时,收缩纤维(retraction fibers, RFs)从细胞后端的质膜中被拉出[1],随后在收缩纤维上出现迁移体。当细胞迁移时,留下的迁移体会被遇到的其他细胞所摄取从而发挥一定的生理功能[2]。据报道,迁移体能调控关键的细胞生理进程,包括线粒体质量控制[3]、细胞间通信[4]和mRNA及蛋白质的横向转移[5]等。先前的研究揭示了迁移体的形成机制。四旋蛋白(Tetraspanin4, TSPAN4)与胆固醇作为迁移体的重要组成部分,其在收缩纤维上的富集导致了迁移体的形成[6]。整合素(Integrin)与细胞外基质组分的正确配对也是迁移体形成的关键因素[7]。另外,高通量筛选已经确定Rho相关的激酶1 (ROCK1)是迁移体形成的调节因子[8]。
值得注意的是,迁移体的形成严格依赖于细胞迁移。使用能干扰细胞迁移的肌球蛋白II抑制剂blebbistatin处理细胞后,可以抑制迁移体的形成[2]。然而,目前不同的细胞迁移模式究竟如何影响迁移体的形成还没有深入探究。
为了去明确不同迁移模式对于迁移体形成的影响,研究人员首先通过活细胞成像,观察了单个TSPAN4-GFP标记的L929细胞的随机迁移,作者观察到当细胞进行非持续性迁移时,例如不同程度的转向,其在转向区域的迁移体数目明显少于在直行区域的数目。为了进一步解释这一现象,研究人员定义了细胞迁移时收缩纤维和迁移体形成相关的形态学指标,包括细胞的形心centroid位置,细胞尾部rear end宽度,收缩纤维主枝parental RFs数量等。随着进一步统计和分析,研究人员最终发现细胞在转向过程中,伴随着迁移细胞被伸长,细胞的尾部变窄,伸出的回缩纤维变少,这就是迁移体在转向的收缩纤维上更少的原因。除了细胞迁移的持续性,细胞迁移的速度也会影响迁移体的形成。更高的迁移速度在同样时间内会导致更长的收缩纤维的产生继而生成更多的迁移体。研究人员在其它两种细胞系(MGC803人结肠癌细胞,NRK大鼠肾细胞)中也验证了这一结论。
研究人员尝试筛选出一种分子不在迁移体上有内源表达,但调控细胞迁移行为,最终他们把目光放到了中间丝(Intermediate filament)波形蛋白(vimentin)上[9]。最近的研究表明,波形蛋白通过调节细胞骨架动力学进而驱动细胞迁移。波形蛋白能通过GEF-H1和RhoA控制肌动蛋白应力纤维[10] 或能调节板状伪足(lamellipodia)的形成来促进细胞迁移[11]。另外,也有报道波形蛋白可以稳定微管网络[12]或强化局部黏附[13]以增强细胞迁移。相比于微丝和微管,波形蛋白并不在回缩纤维和迁移体上有内源分布,即它不可能作为结构组份直接影响迁移体的生物发生。研究人员通过CRISPER/Cas9的方法构建了波形蛋白缺失的L929 TSPAN4-GFP稳定细胞株,首先确定了波形蛋白缺失的确可以导致细胞运动的缺陷,具体表现为细胞迁移速度和迁移持续性的下降,这种下降可以被回补vimentin-mCherry部分恢复。随后,作者验证了波形蛋白的敲除导致了迁移体生成的缺陷也是通过影响了收缩纤维实现的。
在本研究中,作者发现:1)在非持续性迁移时,细胞形成更少的迁移体,原因是细胞在转向时细胞尾部变窄,产生更少的回缩纤维;2)除了运动的持续性,细胞迁移速度通过控制回缩纤维的长度来限制迁移体形成;3)波形蛋白的缺失导致细胞迁移在持续性和迁移速度上均产生了缺陷,相比于野生型细胞产生更少的迁移体。这些结果验证了作者的假设,即迁移体的形成与细胞迁移的持续性和迁移速度直接相关,填补了细胞迁移模式与迁移体形成相关性的研究空白。(图1)
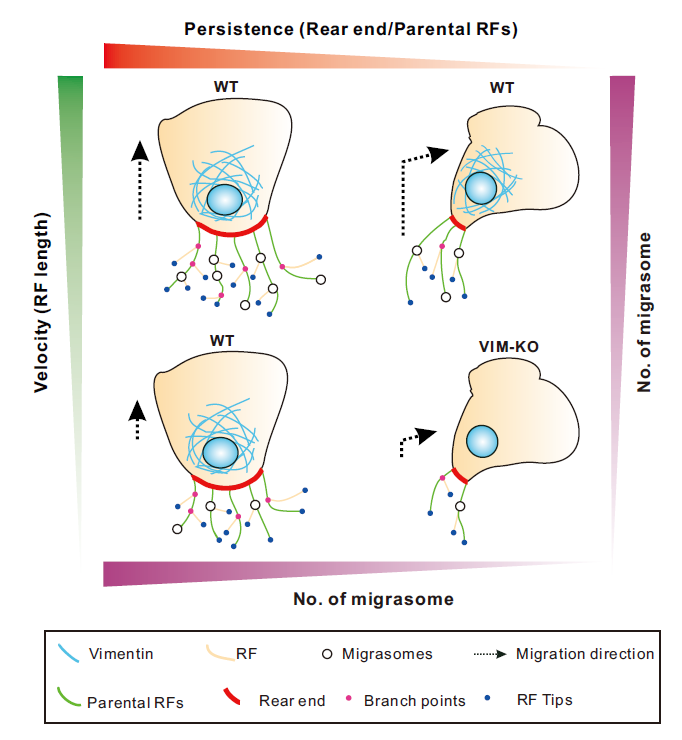
图1.细胞迁移模式影响迁移体形成的示意图。
中国科学院上海巴斯德研究所博士生范昌远为该文章第一作者;中国科学院上海巴斯德研究所酒亚明研究员为该文章的通讯作者;浙江大学季葆华教授和中科院苏州生物医学工程研究所李辉研究员为该文章的数据分析提供了支持;该研究也得到了清华大学俞立教授馈赠细胞等支持和帮助。
【实验室简介】
中国科学院上海巴斯德研究所酒亚明课题组一直围绕细胞骨架调控肿瘤转移和病原体感染这两大类疾病研究,优化并融合了一系列先进光学成像技术和病毒细菌感染实验体系,从细胞骨架、信号转导和病原体宿主互作等多个角度,深入解析了细胞骨架与疾病的分子机理,旨在为癌症以及相关感染性疾病的精准治疗提供新理论、药物靶点和治疗策略。酒亚明研究员围绕上述工作取得一系列重要原创性成果,发表高水平SCI学术论文40余篇,近期研究成果以通讯作者在PNAS, JCB, Current Biology等高水平杂志发表。作为一支朝气蓬勃、高效干练、团结协作的科研团队,课题组热忱欢迎优秀的博士后、博士及研究生联系加入,联系方式ymjiu@ips.ac.cn。
原文链接:https://rupress.org/jcb/article-abstract/221/4/e202109168/213015/Cell-migration-orchestrates-migrasome-formation-by?redirectedFrom=fulltext
参考文献:
[1] TAYLOR, A. C., & ROBBINS, E. (1963). Observations on microextensions from the surface of isolated vertebrate cells. Developmental biology, 6, 660–673. https://doi.org/10.1016/0012-1606(63)90150-7
[2] Ma, L., Li, Y., Peng, J., Wu, D., Zhao, X., Cui, Y., Chen, L., Yan, X., Du, Y., & Yu, L. (2015). Discovery of the migrasome, an organelle mediating release of cytoplasmic contents during cell migration. Cell research, 25(1), 24–38. https://doi.org/10.1038/cr.2014.135
[3] Jiao, H., Jiang, D., Hu, X., Du, W., Ji, L., Yang, Y., Li, X., Sho, T., Wang, X., Li, Y., Wu, Y. T., Wei, Y. H., Hu, X., & Yu, L. (2021). Mitocytosis, a migrasome-mediated mitochondrial quality-control process. Cell, 184(11), 2896–2910.e13. https://doi.org/10.1016/j.cell.2021.04.027
[4] Jiang, D., Jiang, Z., Lu, D., Wang, X., Liang, H., Zhang, J., Meng, Y., Li, Y., Wu, D., Huang, Y., Chen, Y., Deng, H., Wu, Q., Xiong, J., Meng, A., & Yu, L. (2019). Migrasomes provide regional cues for organ morphogenesis during zebrafish gastrulation. Nature cell biology, 21(8), 966–977. https://doi.org/10.1038/s41556-019-0358-6
[5] Zhu, M., Zou, Q., Huang, R., Li, Y., Xing, X., Fang, J., Ma, L., Li, L., Yang, X., & Yu, L. (2021). Lateral transfer of mRNA and protein by migrasomes modifies the recipient cells. Cell research, 31(2), 237–240. https://doi.org/10.1038/s41422-020-00415-3
[6] Huang, Y., Zucker, B., Zhang, S., Elias, S., Zhu, Y., Chen, H., Ding, T., Li, Y., Sun, Y., Lou, J., Kozlov, M. M., & Yu, L. (2019). Migrasome formation is mediated by assembly of micron-scale tetraspanin macrodomains. Nature cell biology, 21(8), 991–1002. https://doi.org/10.1038/s41556-019-0367-5
[7] Wu, D., Xu, Y., Ding, T., Zu, Y., Yang, C., & Yu, L. (2017). Pairing of integrins with ECM proteins determines migrasome formation. Cell research, 27(11), 1397–1400. https://doi.org/10.1038/cr.2017.108
[8] Lu, P., Liu, R., Lu, D., Xu, Y., Yang, X., Jiang, Z., Yang, C., Yu, L., Lei, X., & Chen, Y. (2020). Chemical screening identifies ROCK1 as a regulator of migrasome formation. Cell discovery, 6(1), 51. https://doi.org/10.1038/s41421-020-0179-6
[9] Ivaska, J., Pallari, H. M., Nevo, J., & Eriksson, J. E. (2007). Novel functions of vimentin in cell adhesion, migration, and signaling. Experimental cell research, 313(10), 2050–2062. https://doi.org/10.1016/j.yexcr.2007.03.040
[10] Jiu, Y., Per?nen, J., Schaible, N., Cheng, F., Eriksson, J. E., Krishnan, R., & Lappalainen, P. (2017). Vimentin intermediate filaments control actin stress fiber assembly through GEF-H1 and RhoA. Journal of cell science, 130(5), 892–902. https://doi.org/10.1242/jcs.196881
[11] Helfand, B. T., Mendez, M. G., Murthy, S. N., Shumaker, D. K., Grin, B., Mahammad, S., Aebi, U., Wedig, T., Wu, Y. I., Hahn, K. M., Inagaki, M., Herrmann, H., & Goldman, R. D. (2011). Vimentin organization modulates the formation of lamellipodia. Molecular biology of the cell, 22(8), 1274–1289. https://doi.org/10.1091/mbc.E10-08-0699
[12] Gan, Z., Ding, L., Burckhardt, C. J., Lowery, J., Zaritsky, A., Sitterley, K., Mota, A., Costigliola, N., Starker, C. G., Voytas, D. F., Tytell, J., Goldman, R. D., & Danuser, G. (2016). Vimentin Intermediate Filaments Template Microtubule Networks to Enhance Persistence in Cell Polarity and Directed Migration. Cell systems, 3(3), 252–263.e8. https://doi.org/10.1016/j.cels.2016.08.007
[13] Bhattacharya, R., Gonzalez, A. M., Debiase, P. J., Trejo, H. E., Goldman, R. D., Flitney, F. W., & Jones, J. C. (2009). Recruitment of vimentin to the cell surface by beta3 integrin and plectin mediates adhesion strength. Journal of cell science, 122(Pt 9), 1390–1400. https://doi.org/10.1242/jcs.043042