Nucleic Acids Research | 郝沛研究组推出首个开放使用mRNA药物设计平台
2025年5月19日,中国科学院上海免疫与感染研究所郝沛课题组在国际学术期刊 Nucleic Acids Research 发表了题为 “mRNAdesigner: an integrated web server for optimizing mRNA design and protein translation in eukaryotes” 的合作研究论文,推出了国际首个开放使用的mRNA药物设计平台-mRNAdesigner。该研究成果对新一代mRNA核酸药物的定制化设计和高效开发、推动学术发展和打破产业壁垒有重要意义。

mRNA药物正成为现代生物医药领域的重要发展方向,但当前mRNA的设计面临着跨物种适应性不足、忽略上下游非翻译区对表达的调控作用、免疫原性控制不足等多重挑战。对此,郝沛研究团队创新性地将RNA翻译动力学的研究成果融入设计理念,综合考虑了mRNA序列中的密码子偏好性、GC含量、二级结构对蛋白翻译表达的影响,实现对mRNA结构区域(5’UTR、CDS、3’UTR)的整合优化设计。平台设计的编码区序列可以在设定的GC比例的下,提高密码子自适应指数、优化碱基互补配对模式的同时规避长茎环结构和GC富集区,降低mRNA在细胞中的被天然免疫识别和降解的风险。针对5’UTR区域,平台通过提高核糖体负载(MRL)优化设计,促使翻译起始和延伸更为高效。针对3’UTR区域,通过调控元件注释算法,识别并减少与序列降解有关的调控元件的数量(如:AREs, CUREs),从而能整体提升mRNA稳定性和翻译效率。
通过对序列的模拟与实验评估表明经过mRNAdesigner生成的mRNA序列,相较于其野生型序列,其在密码子适应度、碱基互补配对率、长茎环结构有不同程度的提高,并且在真核细胞表达体系中具有更高的蛋白表达量,为核酸疫苗开发、蛋白药物生产及分子生物学研究提供了有力的工具支持。mRNAdesigner 可从 https://www.biosino.org/mRNAdesigner/ 访问 。
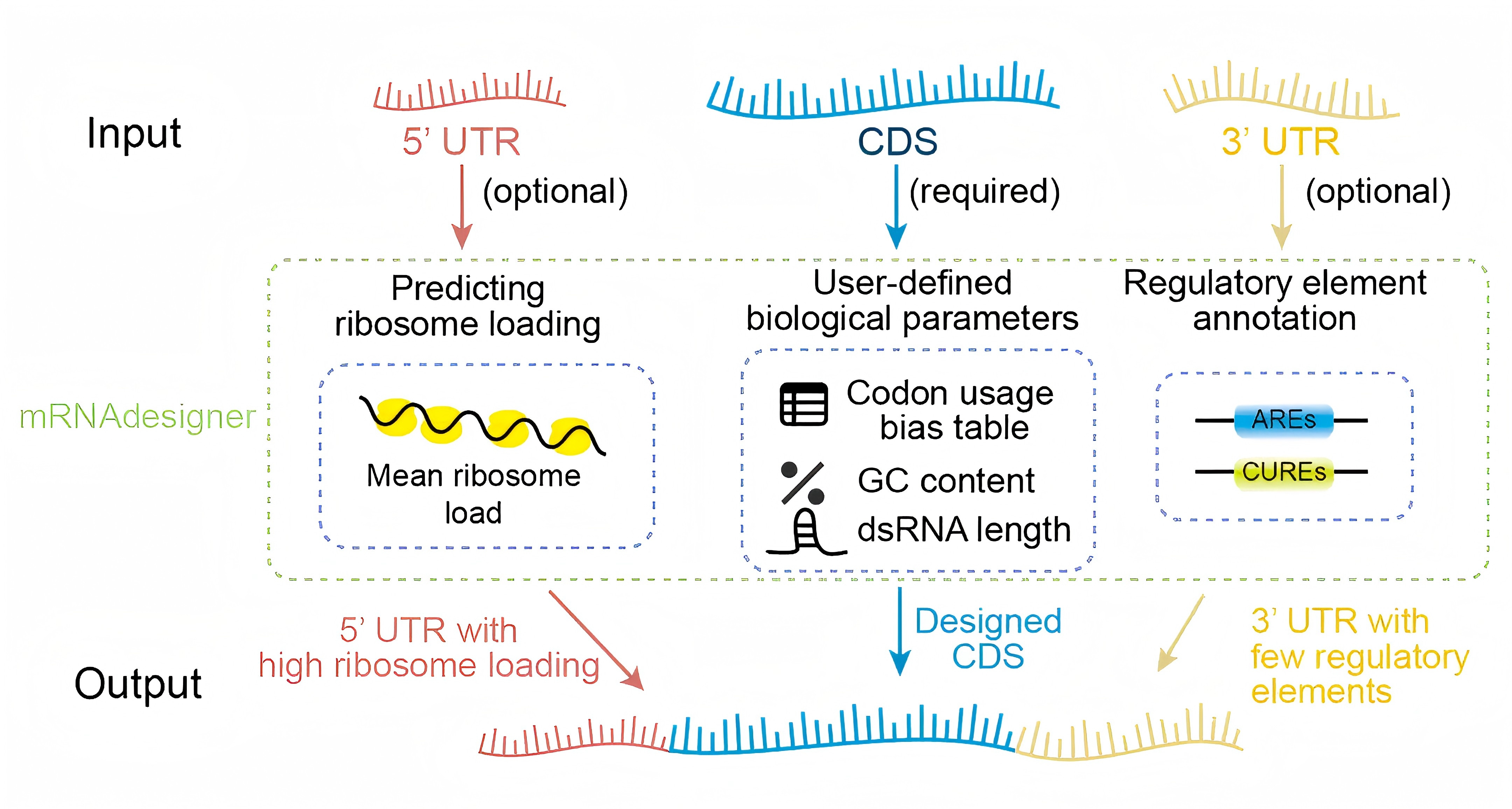
图:mRNAdesigner序列优化流程示意图
原文链接: https://doi.org/10.1093/nar/gkaf410
【研究团队】
中国科学院上海免疫与感染研究所郝沛研究员为该论文的通讯作者,中国科学院分子植物科学卓越中心/合成生物学重点实验室李轩研究员为共同通讯作者。中国科学院上海免疫与感染研究所莫欧阳、张卓,中国科学院分子植物科学卓越新中心程香为论文共同第一作者。华东师范大学药学院、ChemPartner PharmaTech Co.、University of Connecticut的相关老师和同学参与了研究。这项工作得到了非传染性慢性病-国家科技重大专项 [2023ZD0502400] 和国家自然科学基金等项目的支持。
【研究组简介】
病原大数据研究组(负责人:郝沛研究员):主要从事计算生物学/生物信息学领域的交叉前沿研究,充分发挥计算机科学背景,在以下三个方面进行研究:1)在病毒分子进化、致病机制、和耐药性突变规律研究方面,建立关键突变位点的检索算法,病毒致病性的网络模型、和分子动力学模拟评估传播宿主的方法,并进行临床表型关联分析;对SARS病毒和新型H7N9禽流感病毒的分子进化、病毒宿主变迁、和临床诊断分型及治疗具有重大指导意义;2)推广生物信息学方法在病原微生物检测预防和抗药性突变评估的转化应用,建立了一批流行性病毒突变数据库和检索分析工具平台;3)多维组学数据Meta-分析算法探索及应用,开发完成了高通量转录组数据的组装算法的优化模拟、基于组学功能模块的小分子功能表型搜索比对算法、和基因组水平的基因稳定性/敏感性meta-评估算法。相关研究成果发表在Science、Nature、LANCET、PNAS、J BiolChem、BMC Systems Biol、BMC Bioinformatics、Genomics等专业学术杂志上。
附件下载: